ODKRYJ
kluczowe warianty strukturalne
w nowotworach hematologicznych za pomocą Optycznego Mapowania Genomu (OGM)
Strukturalne warianty w nowotworach hematologicznych
Aberracje chromosomalne są głównym czynnikiem biorącym udział w patologii nowotworów hematologicznych. Pierwszym etapem w zrozumieniu etiologii i progresji tych nowotworów jest wykrycie i zidentyfikowanie wariantów strukturalnych i wariantów liczby kopii. Dzięki tej wiedzy można uzyskać:
- Wzrost liczby reprezentatywnych próbek
- Zapewnienie szybszego ustalenia patogenicznych genomów
- Wzrost dokładności stratyfikacji ryzyka związanego z próbką
Optyczne mapowanie genomu odkrywa to, co chcesz zobaczyć
OGM wychwytuje wszystkie klasy wariantów strukturalnych i liczby kopii w jednym teście – detekcji, które zwykle wymagają wielu technik cytogenetycznych. Teraz laboratoria mogą generować bardziej znaczące informacje w prostszy i bardziej wydajny sposób.
Platforma OGM obejmująca cały genom zapewnia cyfrową precyzję, bez szumów, w rozwiązywaniu nierozwiązanych przypadków nowotworów hematologicznych.
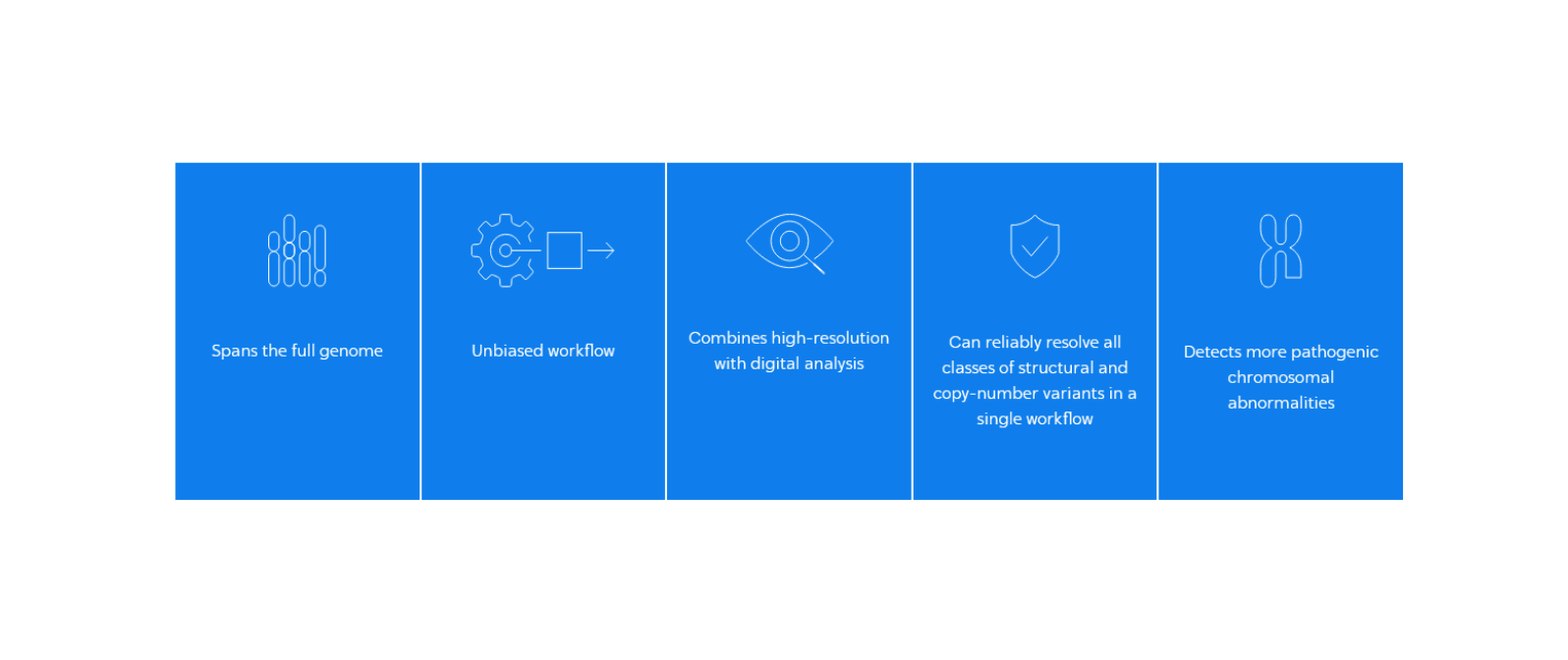
Dowiedz się o zaletach OGM w nowotworach hematologicznych
Następna rewolucja cytogenomiczna w nowotworach hematologicznych
Cytogenomika ma ogromne znaczenie w rozumieniu etiologii i progresji nowotworów hematologicznych. Odkryj zalety innowacyjnej metody OGM na podstawie rzeczywistych, światowych doświadczeń popartych recenzowanymi publikacjami.

OGM rewolucjonizuje sposób, w jaki cytogenetyka przyczynia się do badań nad rakiem
OGM posuwa do przodu dziedzinę badań na nowotworami hematologicznymi w dwojaki sposób:
- Zapewnia pojedynczy test do wykrywania aberracji chromosomalnych od klinicznym i biologicznym znaczeniu, podczas gdy tradycyjne techniki mogą je wykryć dopiero po dwóch, często więcej testach
- Odkrywa więcej dzięki wyższej rozdzielczości, która umożliwia rozróżnienie złożonych struktur, które w przeciwnym razie byłyby tajemnicze lub całkowicie pomijane przez klasyczne techniki.


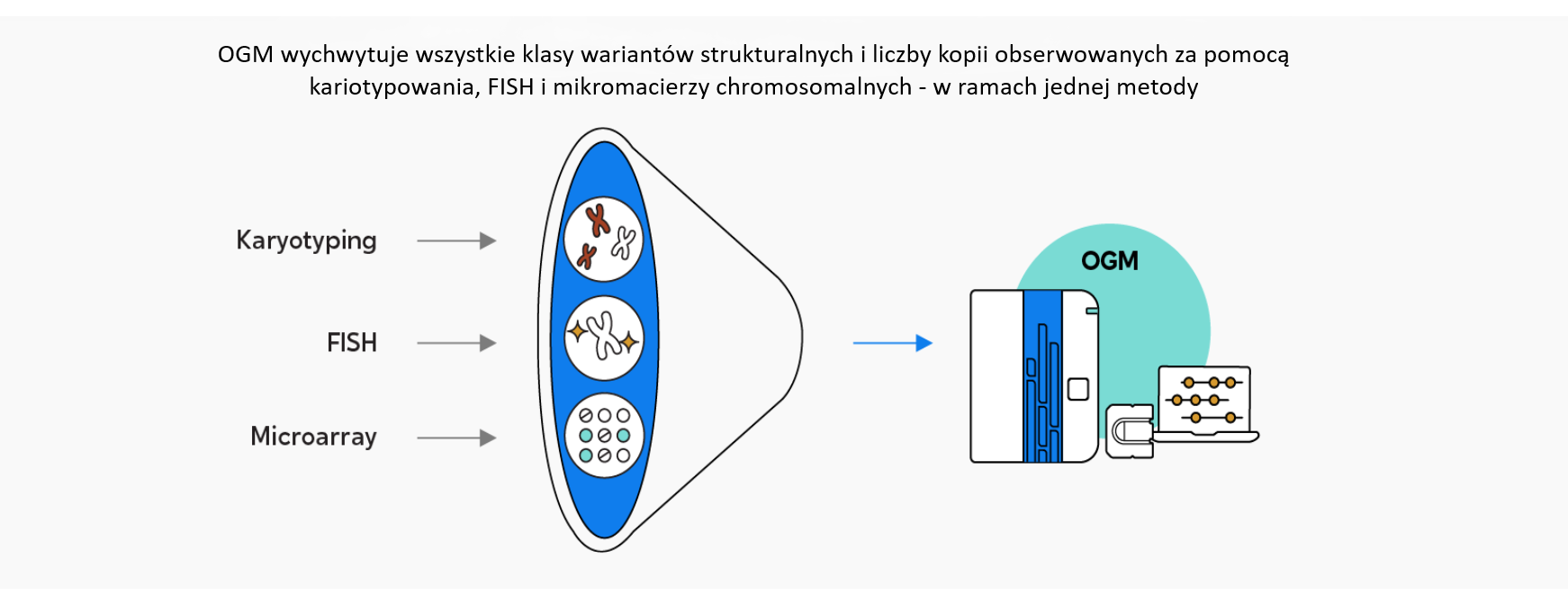
Przełom w odkrywaniu kluczowych spostrzeżeń genomicznych
10,000 razy większa rozdzielczość i wyższa czułość w porównaniu do kariotypowania
Wyniki ponad 99% zgodne ze standardowymi metodami cytogenetycznymi.
5% wartość dla częstość alleli (VAF)
