
TECHNOLOGIA Direct Label and Stain Technology (BEZPOŚREDNIE ZNAKOWANIE I PLAMY)
Technologia Bionano Direct Label and Stain (DLS) jest nową nieinwazyjną chemiczną metodą znakowania motywów sekwencyjnych genomowego DNA, która poprawia każdy aspekt genomowego mapowania w systemie Bionano.
INNOWACJA, KTÓRA SPRAWIA, ŻE WSZYSTKO STAJE SIĘ LEPSZE
Zanim powstało DLS, DNA było oznaczane za pomocą tnących endonukleaz, które wykonywały nacięcia w miejscach charakterystycznych sekwencji, które są oznaczane fluorescencyjnie, a następnie naprawiane. Proces ten jest bardzo trwały oraz specyficzny, jednak, kiedy nacięcia są blisko siebie na przeciwnych niciach DNA, wprowadza systematyczne przerwy, które ograniczają spójność map Bionano.
Metoda DLS zostawia próbkę DNA nienaruszoną, eliminując systematyczne przerwy molekularne. Protokół DLS składa się z pojedynczej enzymatycznej reakcji oznaczania, po której następuje oczyszczanie oraz barwienie. Naprawa nacięć nie jest wymagana, co znacznie usprawnia metodę.
DLS dostarcza najlepsze możliwe mapowanie genomu za zaledwie ułamek ceny.
- Najdłuższe cząsteczki jakie kiedykolwiek uzyskano– Cząsteczki oznaczone za pomocą DLS mogą być znacznie dłuższe, przy często obserwowanych natywnych cząsteczkach >2 Mbp.
- Mapy o długości ramion chromosomów– Mapy genomów stworzone za pomocą DLS są średnio 50-krotnie dłuższe, tworząc lepsze wizualizacje struktury genomu oraz tworząc bardziej przyległe oraz dokładne złożenia. Ramiona chromosomów oraz pełne chromosomy są często składane w pojedyncze mapy.

Mapy zbudowane metodą znakowania opartą na nikazie (NLRS, u góry) obejmują cały ludzki chromosom 3 w celu kompleksowego wykrywania, ale są bardzo fragmentaryczne. W przypadku DLS (na dole) każde ramię chromosomu jest pokryte tylko jedną mapą dla każdego haplotypu.
- Wykrywanie Wariantów Strukturalnych zaczynając od 500 bp- DLS poprawia czułość dla wszystkich wariantów strukturalnych z insercjami oraz delecjami wykrywanymi przy 500 bp.
- Koszt całkowity to $500 dla genomu (ludzkiego, ludzkiego rozmiaru) – DLS sprawia, że mapowanie o dużej objętości staje się bardziej przystępne oraz osiągalne dla użytkowników Bionano.
DLS DO WYKRYWANIA WARIANTÓW STRUKTURALNYCH
DLS jeszcze bardziej usprawnia wiodącą technologię Bionano służącą do wykrywania wariantów strukturalnych oraz zdolność do wyjaśniania skomplikowanych złożeń. Z pojedynczych map na haplotyp, w jednym ramieniu chromosomu, złożone przegrupowania są obrazowane na jednej ciągłej mapie.
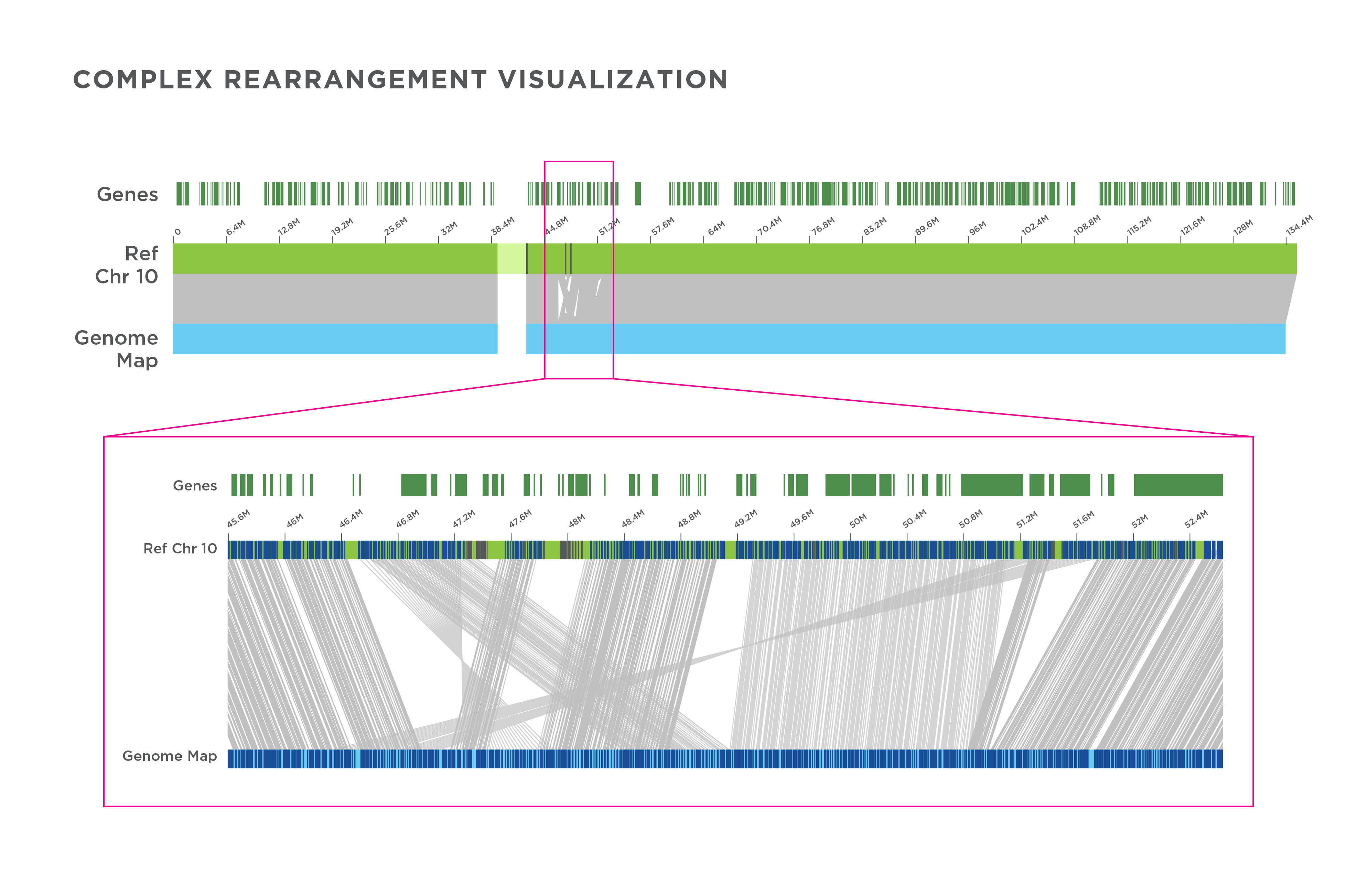
DLS składa pełny ludzki chromosom 10 w dwie pojedyncze mapy, oddzielone centromerem (góra). Pozycje genów przedstawione są jako kolor zielony. Zaznaczenie pokazuje wielokrotne duże przegrupowania złożone w mapę pojedynczą w regionie 10q11.21, związany z niepełnosprawnością intelektualną/ opóźnieniach rozwoju.
Mapy chromosomowe o długości chromosomów oznaczają, że nie ma górnego limitu dla wykrywania wariantów. DLS znacznie poprawia możliwości wykrywania zdarzeń o wielkości kilku milionów par zasad z wysoką czułością. Dodatkowo, nowy algorytm analizy liczby kopii normalizuje pierwotny profil pokrycia w celu dostarczania wywołań liczby kopii wystarczających do wykrywania aneuploidy, utraty ramion chromosomów oraz dużych insercji i delecji powyżej 500 kbp.
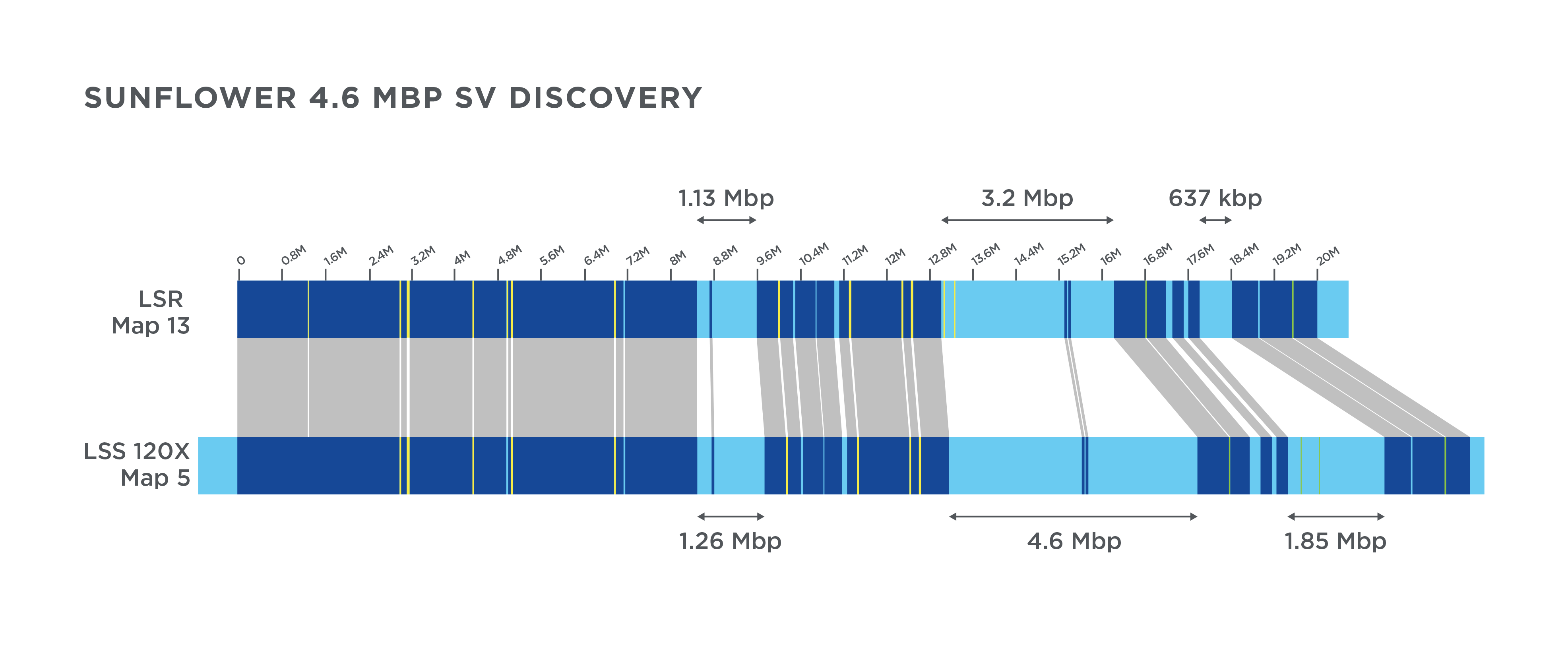
Dwie odmiany słonecznika zostały zmapowane za pomocą DLS oraz złożone de novo w nadzwyczajnie długie mapy. Zidentyfikowano duże warianty strukturalne pomiędzy dwiema odmianami.
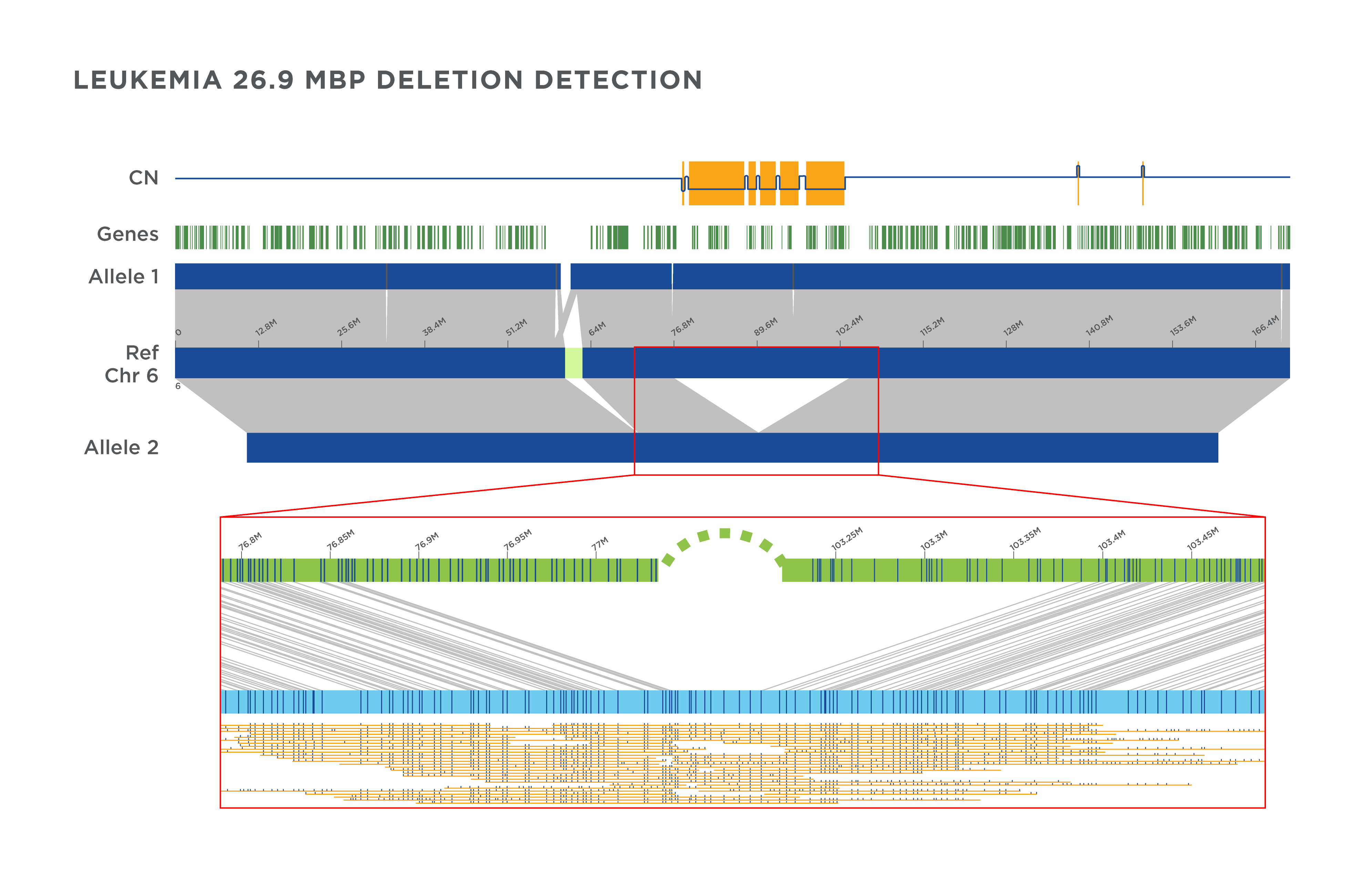
Mapy o długości ramion chromosomów oraz nowe profilowanie liczby kopii (CN) zapewniają solidne narzędzie do wykrywania oraz wizualizacji SV na dużą skalę, tutaj pokazano delecję o wielkości 27 Mbp w genomie pacjenta cierpiącego na białaczkę. Ta sama delecja jest również niezależnie wykrywana z konsensusowej mapie genomu (niebieska mapa przyrównana do referencji w kolorze zielonym). Cząsteczki wyrównane do mapy genomu są przedstawione poniżej.
Poprawiona czułość dla wszystkich wariantów zaczynających się od 500 bp
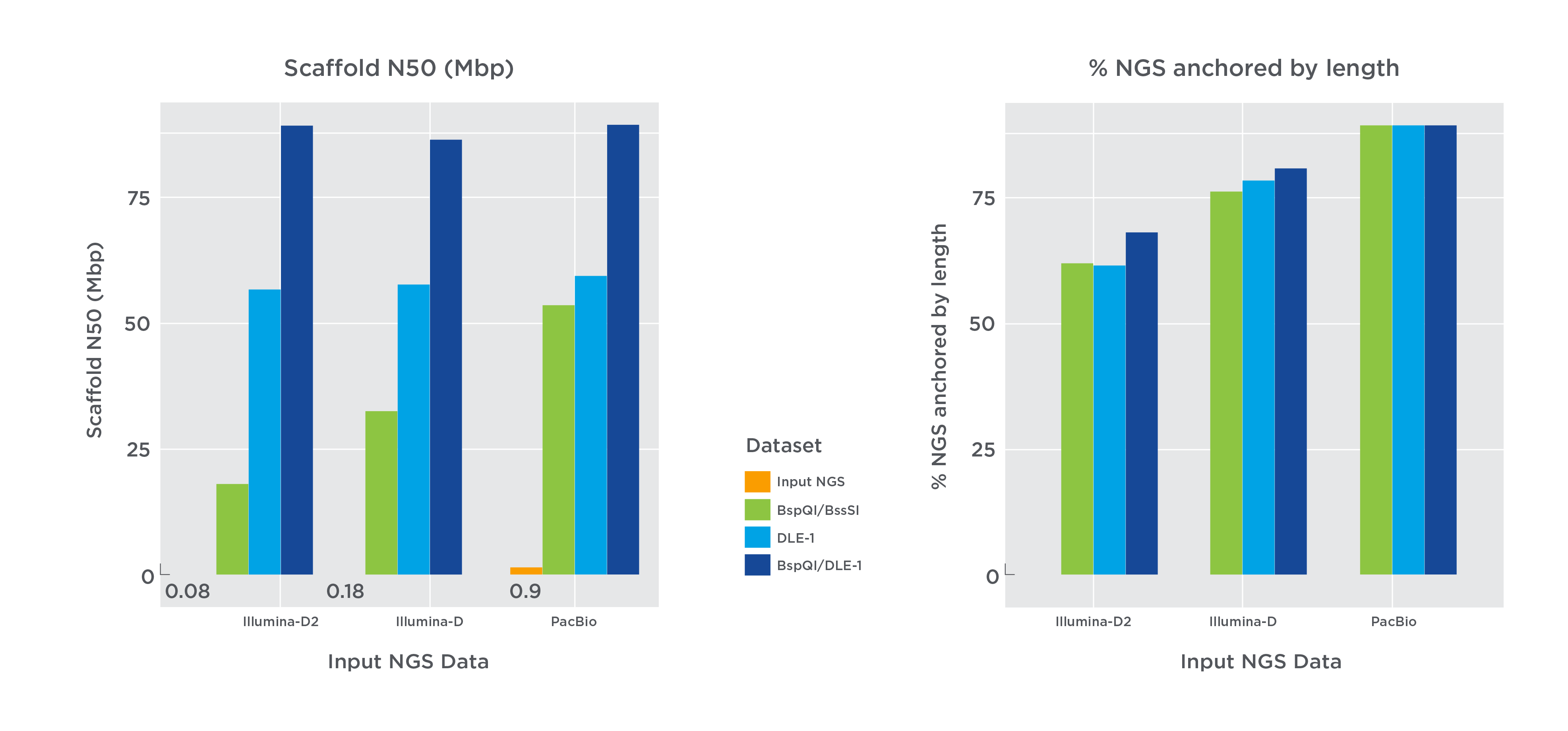
Algorytm Bionano wykrywa wszystkie główne warianty strukturalne (SV) z czułością aż do 99% oraz PPV do ponad 98%. Wykrywanie SV z pojedynczym złożeniem DLS jest równie lub bardziej czułe niż detekcja za pomocą danych z dwóch złożeń stworzonych dzięki nikazie. DLS wykrywa translokacje zaczynając od 500 kbp oraz większe insercje z większą czułością, niż inne poprzednie metody.
DLE-1, pierwszy enzym w rodzinie DLS, posiada zwiększoną gęstość znakowania w genomie człowieka, który jest w stanie wykryć insercje oraz delecje o wielkości 500 bp. Zakres ten przedstawia znaczną poprawę względem minimalnej granicy dla metod opartych na nikazie- 1,000 bp. W porównaniu do insercji oraz delecji wykrywanych w ludzkim genomie przez NGS, wywołania SV w Bionano różnią się jedynie średnio o 60 bp.
Podwójna wydajność. Zmniejszone koszty.
DLS obniża koszt jednego genomu (ludzkiego lub o wielkości genomu ludzkiego) do $500 dla użytkowników korzystających z dużej przepustowości. Wcześniej, uzyskiwanie najwyższej czułości wykrywania SV wymagało mapowania genomu za pomocą dwóch osobnych reakcji znakujących. Teraz wymagana jest tylko jedna reakcja DLS.
Z pomocą DLS, efektywnie można podwoić przepustowość wykrywania SV przez Saphyr, umożliwiając tym samym mapowanie dwóch genomów ludzkich na jednym chipie Saphyr podczas jednej 24-godzinnej sesji.
ZŁOŻENIE GENOMU W TECHNOLOGII DLS
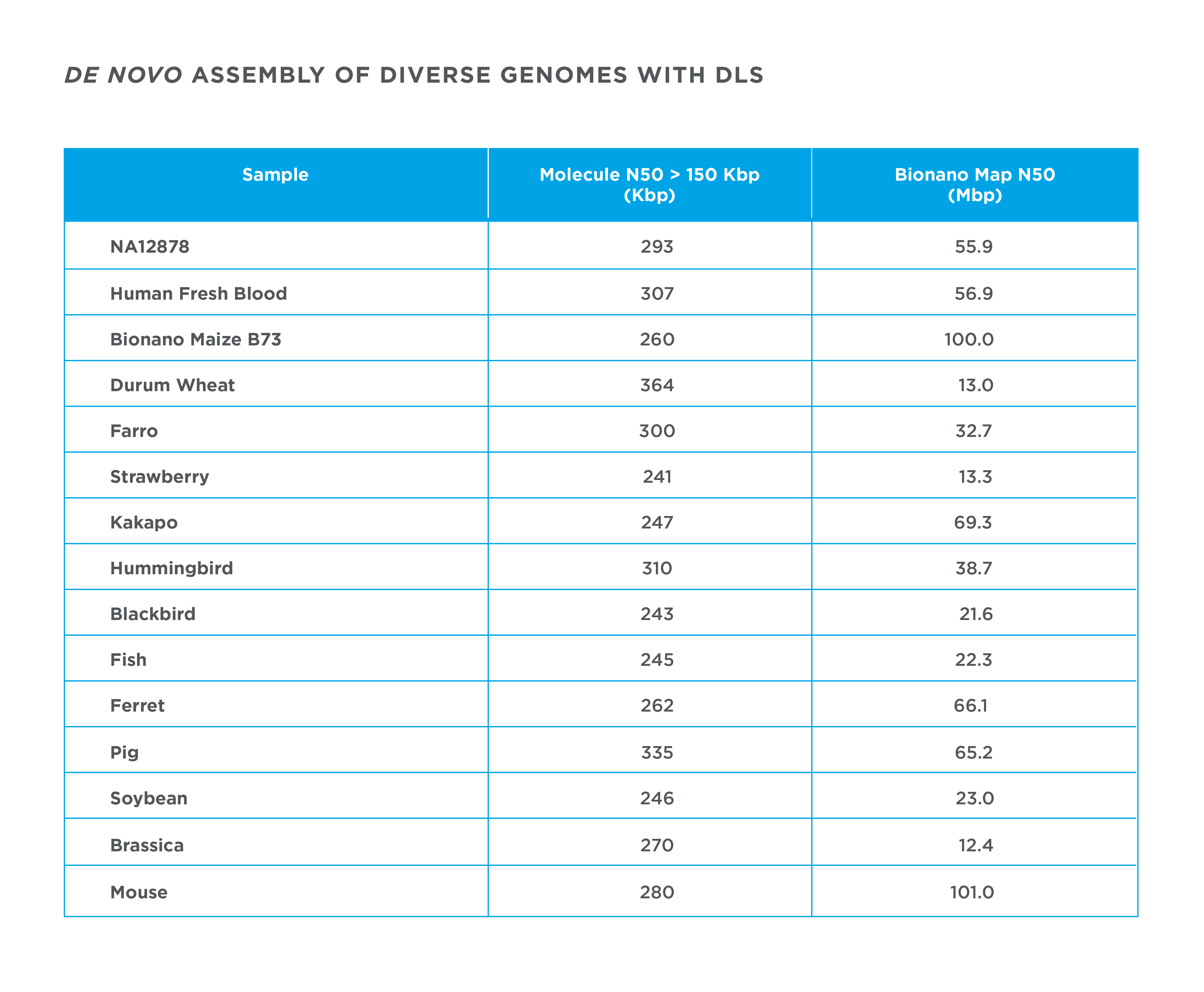
Organizmy złożone de novo za pomocą Bionano Direct Labeling Chemistry (DLS). Złożenia de novo często pokrywają całe ramiona chromosomów, załamując się jedynie na centromerach lub w innych, mniej złożonych miejscach, które są dłuższe od największych zmapowanych cząsteczek.
Ciągłość aż do długości chromosomów
Złożenia de novo pochodzące z genomów oznaczonych przez DLS są jednymi z najbardziej spójnych, zwykle pokrywając pełne ramiona chromosomów. Osiągnęliśmy również pełne rusztowania chromosomowe w niektórych genomach roślinnych oraz zwierzęcych .
Złożenia stworzone z użyciem DLS postawiły poprzeczkę dla spójności oraz dokładności rusztowań, często przewyższając ostatnie publikacje o genomach referencyjnych
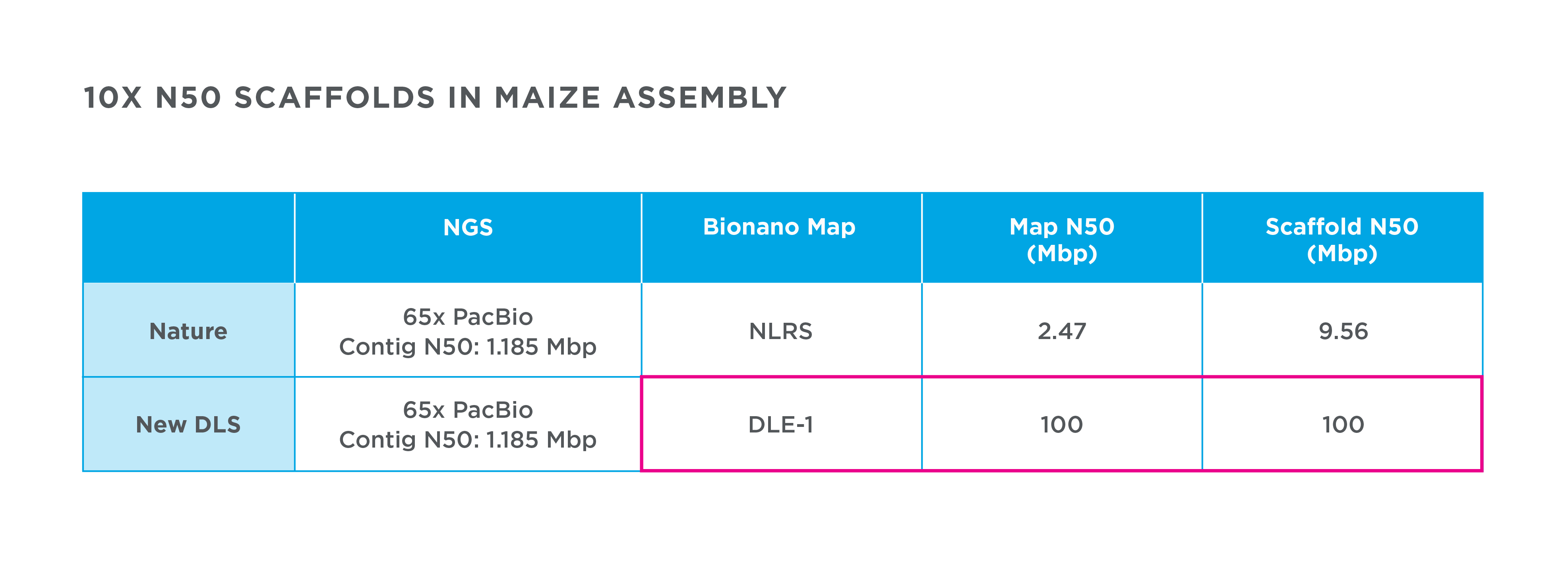
Sekwencja złożenia kukurydzy skafoldowana metodą DLS jest 10-krotnie bardziej przyległa niż niedawno opublikowany w Nature (546), 524-527 (22 Czerwiec 2017) genom referencyjny kukurydzy.
Wykorzystaj w pełni dane sekwencyjne
Posiadając długość ramion chromosomów, mapy Bionano tworzą całkowicie niezależną mapę genomu, który jest wykorzystywany do walidacji oraz korekcji kontigów sekwencyjnych. Chimeryczne kontigi sekwencyjne, powszechne źródło błędów składania sekwencji, zostają wycięte automatycznie.
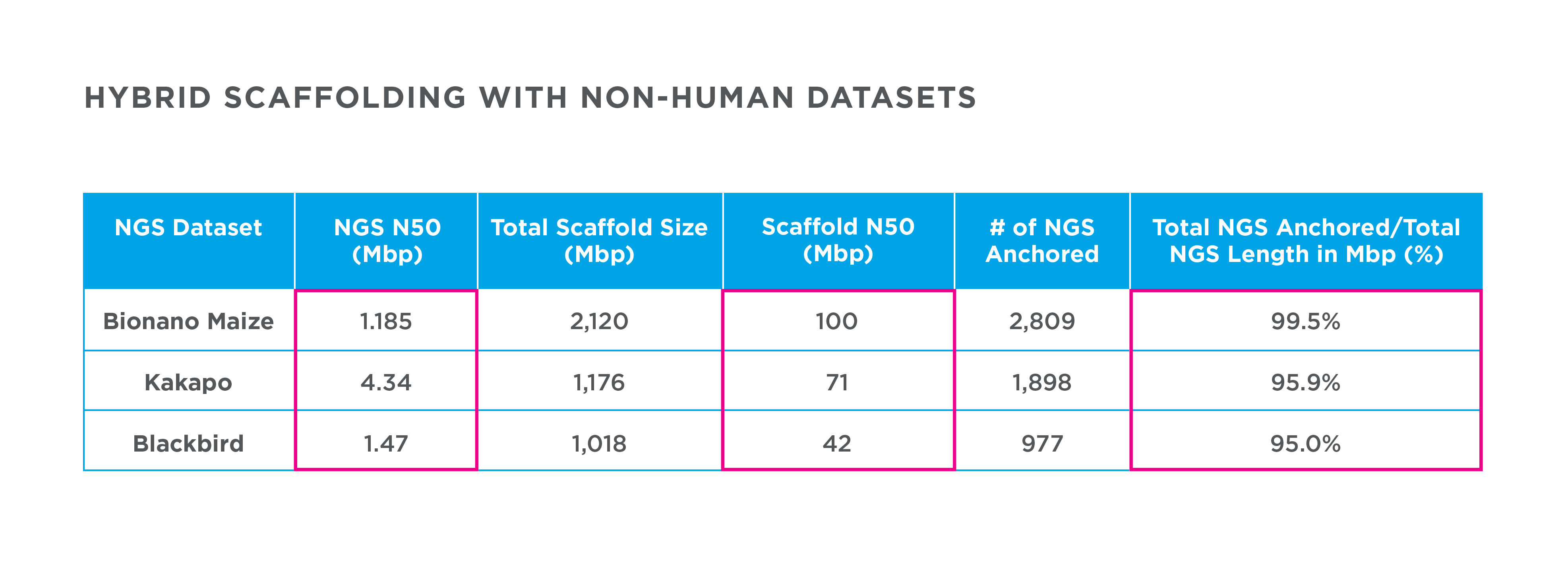
DLS jest kompatybilne ze wszystkimi danymi sekwencji NGS. Wysokiej jakości sekwencje NGS składane z użyciem map DLS dostarczają lepsze wyniki. Dla złożeń o mniejszej przyległości, DLS może zostać połączone ze znakowaniem Bionano NLRS, aby zawrzeć więcej sekwencji w złożeniu, zmniejszając koszty sekwencjonowania oraz poprawiając wyniki.
Wymagania techniczne DLS
DLS jest kompatybilny z DNA izolowanym za pomocą protokołu Bionano Prep.
Bionano Access™ v1.2 oraz Bionano Solve v3.2 zostały zoptymalizowane, aby współdziałały z DLS, z typowymi ludzkimi złożeniami de novo, zajmując mniej niż 30 godzin podczas pojedynczego procesu Saphyr lub usługi Bionano Compute.
DLS nie jest zgodne z systemem Irys®. Skontaktuj się z nami w celu rozszerzenia oferty, jeśli interesuje Cię przejście z Irys na system Saphyr.
